Proteine

Karl-Wilhelm Steinfieber
Biophysik, Eiweisse, Eiweissstoffe, Biomakromoleküle, welche hauptsächlich lineare Polymere der mit Peptidbindungen chemisch verbundenen Aminosäuren sind (Peptidgruppe). Die Bezeichnung »Eiweiss« ist historisch bedingt und geht auf die Isolierung aus Hühnereiweiss zurück. Die Trockensubstanz von Zellen besteht zu über 50 % aus Protein. Proteine können u.a. strukturelle und katalytische (Enzyme) Aufgaben übernehmen. Durch die Wirkung der Proteine wird das genetische Programm, welches in der DNS gespeichert ist, realisiert.
Die Struktur von Proteinen kann mit einer hierarchischen Klassifikation beschrieben werden:
Das unterste Niveau der Strukturorganisation, die Primärstruktur, ist zunächst beim Entstehen des Proteins (Translation) identisch mit der Aminosäuresequenz. Die Reihenfolge der Aminosäuren, wie sie im genetischen Code fixiert ist, kann während der Translation durch Recoding-Mechanismen bzw. durch Protein Splicing und chemische Modifikation der Aminosäureseitenketten (Posttranslationsmodifikation) geändert werden. Zwischen Cysteinresten können sich Disulfidbrücken ausbilden (S-S-Bindungen). Auf den meisten Proteinsequenzpositionen hat die chemische Identität keine kritische Bedeutung. Solange der Aminosäureaustausch nicht mit einem dramatischen Wechsel der physico-chemischen Eigenschaften einhergeht, ist der Effekt für die dreidimensionale Struktur und die Funktion des Proteins klein.
Mit der Sekundärstruktur eines Proteins meint man im allgemeinen Fall die bevorzugten relativen Hauptkettenkonformationen (Konformation, siehe Abb. 1) sequentiell nahegelegener Aminosäurereste infolge lokaler Wechselwirkungen. Historisch ist die Definition sogar noch enger. Danach schliesst die Sekundärstruktur nur repetitive lokale Konformationen entlang der Polypeptidkette (d.h. verschiedene Arten von Helices) ein (siehe Tab.). Die klassischen Sekundärstrukturmotive sind die a-Helix und der b-Strand (von denen mehrere ein b-Faltblatt bilden). Weitere Sekundärstrukturelemente sind die Polyprolin-II-Helix, Turns (kurze Schleifen zwischen Sekundärstrukturelementen) und Loops (lange Schleifen).
Supersekundärstrukturen sind typische Packungen von Sekundärstrukturelementen (z.B. das b-Faltblatt oder das Docking zweier a-Helices mit den Winkeln von -40°, -100° oder 20° zwischen den Helixachsen). Ein Proteinfold bezeichnet das Gerüst der Sekundärstrukturelemente mit repetitiver Hauptkettenstruktur; d.h., die a-Helices und die b-Faltblätter (siehe Abb. 2). Der Begriff Proteintopologie schliesst die räumliche Anordnung aller Sekundärstrukturelemente, auch der Loops, ein.
Die Tertiärstruktur wird durch die relative Position der Atome aller Aminosäurereste der Polypeptidketten beschrieben. Äquivalent zur Darstellung in Koordinatenform ist die Auflistung aller paarweisen Abstände. Ein besonderes Merkmal der Tertiärstruktur sind die sog. nichtlokalen Wechselwirkungen, enge Kontakte sequentiell weit auseinanderliegender Aminosäurereste. Eine Tertiärstruktur kann aus mehreren, lokal besonders dicht gepackten Domänen bestehen. Jede Domäne hat ein eigenes Zentrum aus räumlich nahen, nichtpolaren Aminosäuren, welche durch den hydrophoben Effekt zusammengehalten werden.
Die Quartärstruktur ist aus mehreren, nicht durch Peptidbindungen zusammengehaltenen Untereinheiten (jede enthält eine Polypeptidkette) zusammengesetzt.
Natürliche Proteine bilden unter physiologischen Bedingungen stets eine Tertiärstruktur aus (native Struktur). Die Tertiärstruktur genügt den zwei zentralen Konstruktionsbedingungen: Einerseits sind die Atome der Aminosäuren im Innern des Proteins extrem dicht gepackt, so dass selbst Wassermoleküle nicht dazwischen eindringen können. Andererseits gibt es dramatische Unterschiede in der Polarität der Aminosäurereste im Innern (hauptsächlich apolare Reste) und auf der Proteinoberfläche (viele polare Gruppen). Es ist auch eine Funktion der Sekundärstrukturen im Innern des Proteins, alle polare Gruppen mit Wasserstoffbrücken abzusättigen, weil Interaktionen mit Wassermolekülen dort nicht möglich sind. Die Information über die Tertiärstruktur eines Proteins ist in seiner Aminosäuresequenz gespeichert. Spontane Renaturierung kleiner und mittlerer Proteine nach Denaturierung durch Hitze, Guanidiniumchlorid oder Harnstoff führen zur Wiederherstellung der vorherigen Tertiärstruktur (Faltung). Sehr grosse Proteine benötigen zur Faltung Unterstützung durch Chaperone-Eiweiss.
In lebenden Organismen gibt es Millionen unterschiedliche Proteine (Leben). Die unglaubliche funktionelle Diversität (verglichen mit Nucleinsäuren, Lipiden und Kohlenhydraten) erreichen Proteine durch
1. die hohe Zahl verschiedener Monomere (20 Aminosäuren), welche posttranslational modifiziert werden können,
2. die Inkorporation prosthetischer Gruppen, anorganischer oder kleiner organischer Ionen oder Verbindungen, in ihre Struktur,
3. die Variabilität der dreidimensionalen Struktur (es gibt z.B. globuläre und fibrilläre Proteine),
4. die Unterschiede in der Anzahl der Monomere (von einigen Dutzend bis zu 30 000). Eine Domäne besteht aus 80-200 Aminosäuren; komplexe Funktionen werden auf verschiedene Domänen in Multidomänenproteine aufgeteilt.
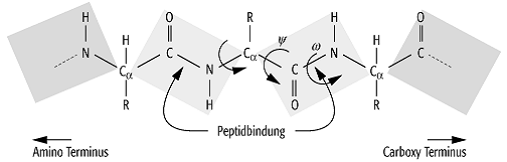
Proteine 1: Die Hauptkette eines Polypeptids. Die Unterteilung
der Hauptkette in Peptideinheiten ist ein geeignetes methodologisches
Herangehen zur Konformationsanalyse, da die Peptidgruppe hauptsächlich planar
ist (der Dihedralwinkel w
definiert durch die Atome Cai-1C\'i-1NiCaI
ist nahe ![]() oder
oder ![]() ). R bezeichnet die
Aminosäureseitenkette. Die Dihedralwinkel f und y, definiert durch die Atome C\'i-1NiCaiC\'I und NiCaiC\'i Ni+1 beschreiben die relativen Lagen der
Peptidgruppen. Im f-y-Diagramm
(Ramachandran-Karte) kann man sterisch erlaubte (infolge der endlichen Radien
der Atome) Konformationen eines Aminosäurerestes innerhalb der Polypeptidkette
illustrieren.
). R bezeichnet die
Aminosäureseitenkette. Die Dihedralwinkel f und y, definiert durch die Atome C\'i-1NiCaiC\'I und NiCaiC\'i Ni+1 beschreiben die relativen Lagen der
Peptidgruppen. Im f-y-Diagramm
(Ramachandran-Karte) kann man sterisch erlaubte (infolge der endlichen Radien
der Atome) Konformationen eines Aminosäurerestes innerhalb der Polypeptidkette
illustrieren.
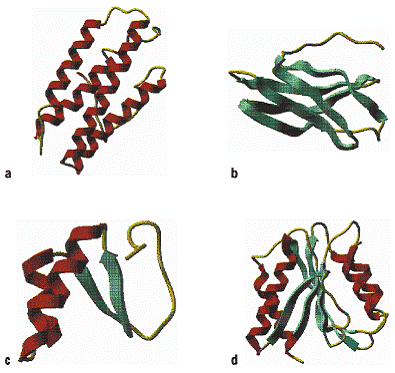
Proteine 2: Beispiele für Proteinfolds: a) rein a-helikales Protein, Myohemerythrin (PDB-Code 2MHR), 4-Helix-Bündel; b) reine b-Faltblattstruktur, Glycosidase Inhibitor (PDB-Code 1HOE), b-Sandwich; c) a + b-Struktur, Crambin (PDB-Code 1CRN); d) a / b-Struktur, Flavodoxin (PDB-Code 2FX2).
Proteine: Typen repetitiver Sekundärstrukturen: Wasserstoffbrücken sind relativ zu einem Rest mit der Sequenzposition i bezeichnet. Während die meisten Helices aus repetitiven trans-Peptidgruppen bestehen, ist Poly-Prolin-I eine Polypeptidekonformation mit cis-Peptidbindungen.
| bottom:none;border-right:none;
padding:0cm 0cm 0cm 0cm\'>
Helixtyp |
bottom:solid black 1.0pt;border-right:none;
padding:0cm 0cm 0cm 0cm\'>
torsion angle [°] |
twist [°] |
pitch [Å] |
|
||
| bottom:solid black 1.0pt;border-right:none;
padding:0cm 0cm 0cm 0cm\'>
|
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
f |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
y |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
w |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
|
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
|
bottom:solid black 1.0pt;border-right:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
|
|
a-Helix |
-57 |
-47 |
180 |
i-i+4 |
100 |
1,50 |
|
310-Helix |
-49 |
-29 |
180 |
i-i+3 |
120 |
2,00 |
|
b-Strand (parallel sheet) |
-119 |
113 |
180 |
- |
180 |
3,2 |
|
b-Strand (antiparallel sheet) |
-139 |
135 |
182 |
- |
180 |
3,4 |
|
Poly-Glycin-II |
-80 |
150 |
180 |
- |
120 |
3,1 |
|
Poly-Prolin-I |
-83 |
158 |
0 |
- |
108 |
1,9 |
| bottom:solid black 1.0pt;border-right:none;
padding:0cm 0cm 0cm 0cm\'>
Poly-Prolin-II |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
-78 |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
149 |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
180 |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
- |
bottom:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
120 |
bottom:solid black 1.0pt;border-right:solid black 1.0pt;
padding:0cm 0cm 0cm 0cm\'>
3,12 |
Techniklexikon.net
Das freie Technik-Lexikon. Fundierte Informationen zu allen Fachgebieten der Ingenieurwissenschaften, für Wissenschaftler, Studenten, Praktiker & alle Interessierten. Professionell dargeboten und kostenlos zugängig.
Techniklexikon
Modernes Studium der Physik sollte allen zugängig gemacht werden.