Profil

Hans-Peter Ahlsen
Allg. Querschnittsform eines Körpers. Stranggepreßte (Extrusion) Rohre und stangenartige Halbzeuge werden als P. bezeichnet. In der Aerodynamik versteht man darunter den Querschnitt von Strömungskörpern, z. B. eines Flugzeugflügels, eines Hubschrauber-Rotorblattes oder einer Turbinenschaufel (Turbine).
Im Modellbau:
parallel zur Strömungsrichtung liegender Schnitt eines Bauteils, mit dem Kräfte in bestimmter Richtung erzeugt werden sollen. P. werden an Modellen vielfältig verwendet, so am Rumpf von Verdrängungsschiffsmodellen, an Rudern, Tragflügeln, Leitwerken, Klappen, Rotorblättern, Propellern u. a. Die Kräfte am Profil greifen am Druckpunkt DP an und werden in die Komponenten Auftriebskraft Fa und Widerstandskraft Fw oder Normalkraft FN und Tangentialkraft FT zerlegt. Die entstehenden Kräfte ändern sich mit der Strömungsgeschwindigkeit und dem Anstellwinkel a. Am P. sind folgende Kennwerte wichtig : Nasenradius r, Profilmittellinie oder Skelettlinie, Sehne (innere, äußere), Wölbung, P. tiefe f und P. dicke D. Die Einhaltung des Nasenradius bei der Herstellung bestimmt die Leistung der P. Die P. dicke wird meist in % von der P. tiefe angegeben. Die P. werden nach der Form unterschieden : 1) symmetrisches P. (Kunstflugmodelle), 2) Profil mit gerader Unterseite (halbsymmetrische P.) (Segelflug- und Motorflugmodelle), 3) unsymmetrisches konvexes P. (Segelflugmodelle, dünne Formen für Freiflugmodelle), 4) gewölbte Platte (einfache Segelflugmodelle), 5) SSchlagP. (Nurflügel modelle). P. werden von aerodynamischen Versuchsanstalten entworfen, in Windkanalmessungen erprobt und in P. tabellen veröffentlicht. Die P. tabellen enthalten die P. koordinaten zum Zeichnen des P. sowie die Beiwerte und den ReZahlbereich. Die P. tabellen oder P. reihen werden mit Kurzzeichen und Ziffern gekennzeichnet. Im Modellbau gebräuchlich sind z. B. WortmannP. (Kurzzeichen FX), NACAP., GöttingerP. (Kurzzeichen Gö), EpplerP. (Kurzzeichen E), Benedek (Kurzzeichen B).
Biophysik, profile, mathematisches
Modell zur Beschreibung der Erwartungswerte für Typen von Aminosäuren in
Sequenzmustern von Biomakromolekülen, insbesondere von Proteinen und zur
quantitativen Bewertung der Kompatibilität (Homologie) einer neuen
Proteinsequenz mit einer bekannten Proteinfamilie (Sequenzanalyse).
Ausgangspunkt ist eine multiples Sequenzalignment einer grossen Anzahl von
Aminosäuresequenzen struktur- bzw. funktionsähnlicher Proteine. Mathematisch
ist das Alignment eine Matrix ![]() , wobei
, wobei ![]() Buchstaben sind, welche einen Aminosäuretyp
bezeichnen, die i-ten Zeilen
Buchstaben sind, welche einen Aminosäuretyp
bezeichnen, die i-ten Zeilen ![]() die Sequenz eines Proteins sind und
die j-ten Spalten
die Sequenz eines Proteins sind und
die j-ten Spalten ![]() die Aminosäuretypen unterschiedlicher Proteine
an der äquivalenten Strukturposition beschreiben. Das Profil ist eine Matrix
die Aminosäuretypen unterschiedlicher Proteine
an der äquivalenten Strukturposition beschreiben. Das Profil ist eine Matrix
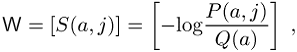
in welcher ![]() die Wahrscheinlichkeit des Auftretens der
Aminosäure a an der Position j
und
die Wahrscheinlichkeit des Auftretens der
Aminosäure a an der Position j
und ![]() die allgemeine
Häufigkeit der Aminosäure a (in bekannten
Proteinen) sind. Es sei
die allgemeine
Häufigkeit der Aminosäure a (in bekannten
Proteinen) sind. Es sei ![]() die
Sequenz eines weiteren Proteins. Die Homologie mit dem Aligment A wird dann durch den Ausdruck S
(Score)
die
Sequenz eines weiteren Proteins. Die Homologie mit dem Aligment A wird dann durch den Ausdruck S
(Score) ![]() bestimmt.
Unter gewissen Bedingungen ist der Score Extremwert-verteilt, so dass eine
Wahrscheinlichkeit der Zugehörigkeit des Proteins b
zur Proteinfamilie A berechnet werden kann.
bestimmt.
Unter gewissen Bedingungen ist der Score Extremwert-verteilt, so dass eine
Wahrscheinlichkeit der Zugehörigkeit des Proteins b
zur Proteinfamilie A berechnet werden kann.
Techniklexikon.net
Das freie Technik-Lexikon. Fundierte Informationen zu allen Fachgebieten der Ingenieurwissenschaften, für Wissenschaftler, Studenten, Praktiker & alle Interessierten. Professionell dargeboten und kostenlos zugängig.
Techniklexikon
Modernes Studium der Physik sollte allen zugängig gemacht werden.